Полиаденилирование (Hklng;yunlnjkfguny)

Полиаденили́рование — это процесс присоединения большого количества остатков аденозинмонофосфата (поли(А)-хвоста) к 3'-концу первичной мРНК (пре-мРНК). Иными словами, поли(А)-хвост — это фрагмент молекулы мРНК, азотистые основания которого представлены только аденином. У эукариот полиаденилирование является частью процессинга мРНК — процесса созревания первичного транскрипта в зрелую мРНК, готовую для трансляции. Процессинг, в свою очередь, является одним из этапов экспрессии генов.
Полиаденилирование начинается, когда завершается транскрипция гена, то есть образование первичного транскрипта. Перед началом полиаденилирования особый мультисубъединичный белковый комплекс отщепляет 3'-концевой участок первичного транскрипта. Место расщепления определяется положением универсальных сигнальных последовательностей в первичном транскрипте; в некоторых случаях расщепление может происходить в нескольких альтернативных сайтах. Таким образом, полиаденилирование даёт возможность для образования различных мРНК одного гена (альтернативное полиаденилирование), подобно тому, как это происходит при альтернативном сплайсинге. После формирования нового 3'-конца транскрипта компонент белкового комплекса поли(А)-полимераза осуществляет синтез поли(А)-хвоста, используя 3'-концевой нуклеотид как затравку[1].
Поли(А)-хвост играет важную роль в транспорте мРНК из ядра, её трансляции и стабильности. Со временем поли(А)-хвост укорачивается, и, когда его длина станет достаточно малой, мРНК разрушается под действием специальных ферментов[2]. Однако в клетках некоторых типов мРНК с короткими поли(А)-хвостами запасаются в цитозоле для дальнейшей активации путём реполиаденилирования[3]. У бактерий, напротив, полиаденилирование запускает разрушение транскрипта[4]. Подобный эффект полиаденилирования отмечен и для некоторых эукариотических некодирующих РНК[5].
Ядерное полиаденилирование
[править | править код]Функции
[править | править код]При ядерном полиаденилировании поли(А)-хвост присоединяется к мРНК в конце транскрипции. Полиаденилирование защищает мРНК от ферментативного разрушения в цитоплазме, способствует терминации транскрипции, участвует в экспорте мРНК из ядра и трансляции[2]. Практически все эукариотические мРНК полиаденилируются[6], исключением являются мРНК гистонов, образование которых зависит от циклов репликации исходной ДНК[7]. Они являются единственными эукариотическими мРНК, у которых отсутствует поли(А)-хвост, вместо него на 3'-конце транскрипта располагается шпилька, за которой находится обогащённая пуринами последовательность (гистоновый нисходящий элемент (англ. histone downstream element)), отмечающая место, где был произведён разрез исходного транскрипта[8].
Многие эукариотические некодирующие РНК также полиаденилируются в конце трансляции. Среди них есть малые РНК, у которых поли(А)-хвост имеется лишь на промежуточной стадии, но удаляется в ходе процессинга и отсутствует у зрелых молекул (таковы, например, микроРНК)[9][10]. Однако у многих длинных некодирующих РНК, по-видимому, являющихся обширной группой регуляторных РНК (например, у РНК Xist, задействованной в инактивации Х-хромосомы), поли(А)-хвост является частью зрелой РНК[11].
Механизм
[править | править код]| Задействованные белки:[6] CPSF[англ.]: фактор специфичности разрезания и полиаденилирования РНК |
В ядре аппарат полиаденилирования работает с продуктами активности РНК-полимеразы II, например, предшественниками мРНК[англ.]. При этом мультибелковый комплекс (см. справа) отрезает наиболее близкую к 3'-концу часть транскрипта и полиаденилирует образовавшийся в результате разреза конец. Этот разрез катализируется ферментом CPSF[англ.][7] и происходит на 10—30 нуклеотидов ниже его сайта связывания[12]. Обычно сайтом связывания CPSF служит последовательность AAUAAA, однако возможны и другие последовательности, с которыми CPSF связывается более слабо[13]. Специфичность связывания с РНК обеспечивается двумя другими белками: CstF и CFI. CstF связывается с GU-богатым участком РНК ниже сайта связывания CPSF[14]. CFI связывается со специальным сайтом на РНК (у млекопитающих это набор последовательностей UGUAA[15][16][17]) и может обеспечить связывание CPSF с транскриптом даже в отсутствие сигнала AAUAAA[18][19]. Сигнал полиаденилирования — особая последовательность, распознаваемая белковым комплексом, разрезающим РНК — варьирует у различных групп эукариот. У человека сигналом полиаденилирования в большинстве случаев служит последовательность AAUAAA[14], но у растений и грибов она является сигналом полиаденилирования реже[20].
Обычно разрезание РНК происходит до завершения транскрипции, поскольку CstF также связывается с РНК-полимеразой II[21]. CstF служит сигналом к диссоциации от цепи для РНК-полимеразы II, однако механизм этого сигнала плохо понятен[22]. В разрезании также принимает участие белок CFII, однако его роль ещё не ясна[23]. Сайт разрезания связан с сигналом полиаденилирования, и его длина может составлять до 50 нуклеотидов[24].
После разрезания РНК начинается полиаденилирование, катализируемое ферментом полиаденилат-полимеразой[англ.] (поли(А)-полимеразой). Поли(А)-полимераза осуществляет наращивание поли(А)-хвоста, добавляя к РНК АМФ, получаемый из АТФ с высвобождением пирофосфата[25]. Другой белок — PAB2 — связывается с новым, ещё коротким поли(А)-хвостом и увеличивает сродство поли(А)-полимеразы к РНК. Когда длина поли(А)-хвоста достигает приблизительно 250 нуклеотидов, поли(А)-полимераза более не может быть связанной с CPSF и полиаденилирование останавливается, тем самым детерминируя длину поли(А)-хвоста[26][27]. Поскольку CPSF также связан с РНК-полимеразой II, он посылает ей сигнал о прекращении транскрипции[28][29]. Когда РНК-полимераза II достигает терминирующей последовательности (ТТАТТ на ДНК-матрице и AAUAAA на первичном транскрипте), происходит терминация транскрипции[30]. Аппарат полиаденилирования также физически связан со сплайсосомой — комплексом, вырезающим интроны из РНК[19].
Последующие эффекты
[править | править код]
Поли(А)-хвост выступает сайтом связывания для поли(А)-связывающего белка[англ.] (PABP). PABP способствует экспорту РНК из ядра и трансляции, в то же время подавляя её деградацию[31]. Связывание этого белка с поли(А)-хвостом происходит до экспорта РНК из ядра. У дрожжей PABP привлекает к транскрипту поли(А)-нуклеазу — фермент, укорачивающий поли(А)-хвост и тем самым делающий возможным транспорт РНК из ядра. Вместе с РНК в цитоплазму перемещается и РАВР. мРНК, не экспортированные в цитоплазму, разрушаются специальным комплексом — экзосомой[32][33]. РАВР также способен связываться и привлекать к транскрипту ряд белков, оказывающих влияние на трансляцию[32], например, фактор инициации[англ.] 4G, который, в свою очередь, участвует в привлечении к мРНК рибосомной субъединицы 40S[34]. Кроме того, PABP за счёт связывания с факторами инициации трансляции, связанными с 5'-концом мРНК, обеспечивает образование замкнутой петли из линейной мРНК (циркуляризацию мРНК). По-видимому, циркуляризация мРНК обеспечивает эффективную «круговую» трансляцию, при которой за счёт сближенности 5'- и 3'-концов рибосома, подошедшая к 3'-концу мРНК, не диссоциирует от неё, а сразу переходит на 5'-конец и начинает новый раунд трансляции[35]. Наконец, кроме общих механизмов воздействия PABP на трансляцию, они могут специфически влиять на трансляцию отдельных мРНК[36]. Тем не менее, поли(А)-хвост не является необходимым для трансляции всех мРНК[37].
Деаденилирование
[править | править код]В соматических клетках эукариот поли(А)-хвост в цитоплазме укорачивается, и мРНК с укороченными поли(А)-хвостами меньше транслируются и быстрее деградируются[38]. Однако до полной деградации мРНК может пройти несколько часов[39]. Это деаденилирование и деградация мРНК могут ускоряться микроРНК, связывающимися с 3'-нетранслируемой областью (3'-UTR) транскрипта[40]. В ооцитах мРНК с укороченными поли(А)-хвостами не разрушаются, а запасаются в неактивном виде без трансляции и впоследствии активируются путём цитоплазматического полиаденилирования, происходящего после оплодотворения в ходе активации яйца[англ.][41]. Это явление получило название «маскирование мРНК»[42].
У животных поли(А)-рибонуклеаза[англ.] может связываться с кэпом и при этом удалять нуклеотиды с поли(А)-хвоста. Доступность связывания с кэпом и поли(А)-хвостом важна для регуляции того, как скоро мРНК будет разрушена. PARN имеет меньшую деаденилирующую активность, если с РНК связаны факторы инициации трансляции 4E в области кэпа и 4G на поли(А)-хвосте, поэтому трансляция сокращает деаденилирование. Скорость деаденилирования может также регулироваться РНК-связывающими белками[англ.]. После удаления с транскрипта поли(А)-хвоста декэпирующий комплекс убирает кэп, что ведёт к деградации РНК. У дрожжей также было идентифицировано несколько других ферментов, задействованных в деаденилировании[43].
Альтернативное полиаденилирование
[править | править код]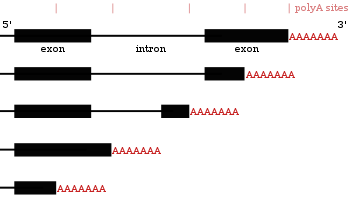
Многие белоккодирующие гены могут иметь более одного сайта полиаденилирования, поэтому с одного и того же гена можно получить несколько РНК, различающихся своими 3'-концами[20][44][45]. Это явление получило название альтернативного полиаденилирования. Поскольку альтернативное полиаденилирование изменяет длину 3'-UTR, оно может повлиять на то, какие сайты связывания с микроРНК останутся на транскрипте[12][46]. Обычно микроРНК подавляют трансляцию и запускают деградацию мРНК, с которыми они связаны, хотя известны примеры, когда микроРНК стабилизирует транскрипт[47][48]. Альтернативное полиаденилирование может также изменить длину кодирующей области, из-за чего получающиеся мРНК будут кодировать различные белки[49][50], однако такое явление встречается реже, чем укорочение 3'-UTR[20].
Выбор сайта полиаденилирования может зависеть от внеклеточных стимулов и экспрессии определённых белков, участвующих в полиаденилировании[51][52]. Например, экспрессия белка CSTF2[англ.] — субъединицы CstF — активирует макрофаги в ответ на липополисахариды (группа бактериальных соединений, вызывающих иммунный ответ). Это приводит к выбору более слабого сайта полиаденилирования и образованию более коротких транскриптов с укороченной 3'-UTR у генов, белковые продукты которых участвуют в осуществлении защитной реакции (например, лизоцима и TNF-α). В результате в этих транскриптах отсутствуют некоторые регуляторные элементы, локализующиеся в 3'-UTR, что увеличивает их срок жизни и даёт возможность для образования большего количества защитных белков[51]. Роль в выборе сайта полиаденилирования могут играть и белки, не связанные непосредственно с аппаратом полиаденилирования[52][53][54][55], например, усиливающие метилирование ДНК рядом с сайтом полиаденилирования[56].
Цитоплазматическое полиаденилирование
[править | править код]В некоторых клетках животных, а именно клетках зародышевой линии в ходе раннего эмбриогенеза, а также постсинаптических участках нейронов происходит полиаденилирование в цитозоле. В ходе цитоплазматического полиаденилирования происходит удлинение поли(А)-хвоста у инактивированных мРНК с укороченным поли(А)-хвостом. В результате цитоплазматического полиаденилирования они активируются и транслируются[38][57]. До этого длина поли(А)-хвоста таких мРНК составляет около 20 нуклеотидов, а в ходе цитоплазматического полиаденилирования он удлиняется до 80—150 нуклеотидов[3].
В раннем эмбрионе мыши цитоплазматическое полиаденилирование инактивированных материнских мРНК, содержавшихся в яйцеклетке до оплодотворения, позволяет клеткам выжить и расти, хотя транскрипция в самом зародыше начинается на двуклеточной (у человека — четырёхклеточной) стадии[58][59]. В мозге цитоплазматическое полиаденилирование активируется в процессе обучения и может играть роль в долговременной потенциации[3][60].
В цитоплазматическом полиаденилировании задействованы РНК-связывающие белки CPSF и CPEB, кроме того, в нём могут и участвовать другие РНК-связывающие белки, например, PUM1[англ.][61]. В зависимости от типа клеток цитоплазматическое полиаденилирование может осуществлять либо поли(А)-полимераза, идентичная той, что задействована в ядерном полиаденилировании, либо цитоплазматическая полимераза GLD-2[62].
Роль в деградации РНК у эукариот
[править | править код]Для многих некодирующих РНК, в том числе тРНК, рРНК, малых ядерных РНК и малых ядрышковых РНК полиаденилирование является меткой для их деградации, по крайней мере, у дрожжей[63]. Полиаденилирование таких РНК осуществляет комплекс TRAMP, присоединяющий около 4 нуклеотидов к их 3'-концу[64]. Меченная таким образом РНК разрушается экзосомой[65]. У человеческих рРНК также были выявлены поли(А)-хвосты, причём среди них были как гомополимерные (состоящие только из А), так и гетерополимерные (состоящие по большей части из А) хвосты[66].
Полиаденилирование у прокариот и органелл
[править | править код]
У многих бактерий полиаденилируются как мРНК, так и некодирующие РНК. Поли(А)-хвосты в этом случае стимулируют деградацию этих РНК специальным мультибелковым комплексом — деградосомой, который включает два РНК-разрушающих фермента: полинуклеотидфосфорилазу[англ.] и РНКазу E[англ.]. Полинуклеотидфосфорилаза связывается с 3'-концом РНК, а поли(А)-хвост за счёт дополнительного места для посадки фермента даёт возможность этому ферменту связаться с РНК, чья вторичная структура делала невозможной посадку на непосредственно 3'-конец. Последующие циклы полиаденилирования и деградации 3'-конца, осуществляемые полинуклеотидфосфорилазой, позволяют деградосоме преодолевать неудобную вторичную структуру транскрипта. Поли(А)-хвост может также привлекать РНКазы, разрезающие РНК на два фрагмента[67]. Такие бактериальные поли(А)-хвосты имеют длину около 30 нуклеотидов[68].
У трипаносом в митохондриях открыты примеры полиаденилирования, как стабилизирующие, так и дестабилизирующие РНК. Дестабилизирующие поли(А)-хвосты известны и для мРНК, и для некодирующих РНК. Средняя длина поли(А)-хвостов в митохондриях трипаносом составляет около 43 нуклеотидов. Стабилизирующие поли(А)-хвосты начинаются со стоп-кодона, и без поли(А)-хвоста в мРНК нет стоп-кодона UAA, потому что в мРНК без поли(А)-хвоста встречаются U и сочетание UA, но не UAA. Для митохондрий растений известно только дестабилизирующее полиаденилирование, а в митохондриях дрожжей полиаденилирования вообще нет[69].
Хотя у бактерий и митохондрий имеется поли(А)-полимераза, у них встречается и другой тип полиаденилирования, осуществляемый полинуклеотидфосфорилазой самой по себе. Этот фермент имеется у бактерий[70], митохондрий[71], пластид[72], а также входит в состав экзосом архей[73]. Он способен синтезировать продолжение 3'-конца, и в этом продолжении подавляющее число азотистых оснований представлено аденином. Как и у бактерий, полиаденилирование, осуществляемое полинуклеотидфосфорилазой, стимулирует разрушение РНК у пластид[74] и, возможно, архей[69].
Эволюция
[править | править код]Хотя полиаденилирование имеется у практически всех организмов, его механизмы не универсальны[75][76]. Однако широкое распространение полиаденилирования и факт того, что оно имеется у организмов из всех трёх доменов жизни, даёт основания предполагать, что последний универсальный общий предок всех организмов в некотором виде имел систему полиаденилирования[68]. Небольшое число организмов не полиаденилируют свои мРНК, что свидетельствует о том, что они утратили способность к полиаденилированию в ходе эволюции. Хотя примеры эукариот, лишённых полиаденилирования, неизвестны, у бактерии Mycoplasma gallisepticum[англ.] и галофильной археи Haloferax volcanii эта модификация отсутствует[77][78].
Наиболее древним полиаденилирующим ферментом является полинуклеотидфосфорилаза. Этот фермент входит в состав бактериальных деградосом и экзосом архей[79] — двух близкородственных комплексов, расщепляющих РНК на нуклеотиды. Этот фермент разрушает РНК, атакуя по фосфату связь между двумя самыми ближними к 3'-концу нуклеотидами, отрывая от РНК дифосфатный нуклеотид. Эта реакция обратима, поэтому этот фермент может и удлинить 3'-конец. Гетерополимерный хвост, добавляемый полинуклеотидфосфорилазой, чрезвычайно насыщен аденином. Выбор из всех азотистых оснований именно аденина для этих целей обусловлен, по-видимому, большей, по сравнению с другими нуклеотидами, концентрацией АДФ, потому что АДФ образуется при расщеплении АТФ для получения энергии; по-видимому, именно это и стало причиной образования поли(А)-хвоста у ранних форм жизни. Предполагается, что участие поли(А)-хвостов в деградации РНК послужило толчком для дальнейшей эволюции поли(А)-полимераз, которые обеспечивают присоединение поли(А)-хвоста, все азотистые основания которого представлены аденином[80].
Поли(А)-полимеразы не являются такими древними ферментами, как полинуклеотидфосфорилаза. У бактерий и эукариот они появились независимо от САА-добавляющего фермента[англ.] — фермента, осуществляющего созревание 3'-концов тРНК. Его каталитический домен не гомологичен таковому у других полимераз[65]. Предполагается, что горизонтальный перенос бактериального САА-добавляющего фермента к эукариотам позволил архееподобному САА-добавляющему ферменту сменить функцию на поли(А)-полимеразную[68]. У некоторых групп организмов, например, архей и цианобактерий, в ходе эволюции поли(А)-полимераза никогда не появлялась[80].
История изучения
[править | править код]Впервые полиаденилирование было идентифицировано в 1960-х как ферментативная активность в экстрактах клеточных ядер, полимеризовавшая АДФ, но не АТФ, в полиаденин[81][82]. Хотя впоследствии такая ферментативная активность была обнаружена в клетках многих типов, её функции были неизвестны до 1971 года, когда поли(А)-последовательности были выявлены в мРНК[83][84]. Первоначально единственной функцией этих последовательностей считали защиту мРНК от действия нуклеаз, в дальнейшем были установлены роли полиаденилирования в транспорте мРНК из ядра и трансляции. Полимеразы, осуществляющие полиаденилирование, были выделены и охарактеризованы в 1960-х и 1970-х, но большое число дополнительных белков, участвующих в этом процессе, было открыто лишь в начале 1990-х[83].
Примечания
[править | править код]- ↑ Proudfoot N. J., Furger A., Dye M. J. Integrating mRNA processing with transcription. (англ.) // Cell. — 2002. — Vol. 108, no. 4. — P. 501—512. — PMID 11909521.
- ↑ 1 2 Guhaniyogi J., Brewer G. Regulation of mRNA stability in mammalian cells. (англ.) // Gene. — 2001. — Vol. 265, no. 1-2. — P. 11—23. — PMID 11255003.
- ↑ 1 2 3 Richter J. D. Cytoplasmic polyadenylation in development and beyond. (англ.) // Microbiology and molecular biology reviews : MMBR. — 1999. — Vol. 63, no. 2. — P. 446—456. — PMID 10357857.
- ↑ Steege D. A. Emerging features of mRNA decay in bacteria. (англ.) // RNA (New York, N.Y.). — 2000. — Vol. 6, no. 8. — P. 1079—1090. — PMID 10943888.
- ↑ Anderson J. T. RNA turnover: unexpected consequences of being tailed. (англ.) // Current biology : CB. — 2005. — Vol. 15, no. 16. — P. 635—638. — doi:10.1016/j.cub.2005.08.002. — PMID 16111937.
- ↑ 1 2 Hunt A. G., Xu R., Addepalli B., Rao S., Forbes K. P., Meeks L. R., Xing D., Mo M., Zhao H., Bandyopadhyay A., Dampanaboina L., Marion A., Von Lanken C., Li Q. Q. Arabidopsis mRNA polyadenylation machinery: comprehensive analysis of protein-protein interactions and gene expression profiling. (англ.) // BMC genomics. — 2008. — Vol. 9. — P. 220. — doi:10.1186/1471-2164-9-220. — PMID 18479511.
- ↑ 1 2 Dávila L. M., Samuelsson T. Early evolution of histone mRNA 3' end processing. (англ.) // RNA (New York, N.Y.). — 2008. — Vol. 14, no. 1. — P. 1—10. — doi:10.1261/rna.782308. — PMID 17998288.
- ↑ Marzluff W. F., Gongidi P., Woods K. R., Jin J., Maltais L. J. The human and mouse replication-dependent histone genes. (англ.) // Genomics. — 2002. — Vol. 80, no. 5. — P. 487—498. — PMID 12408966.
- ↑ Saini H. K., Griffiths-Jones S., Enright A. J. Genomic analysis of human microRNA transcripts. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2007. — Vol. 104, no. 45. — P. 17719—17724. — doi:10.1073/pnas.0703890104. — PMID 17965236.
- ↑ Yoshikawa M., Peragine A., Park M. Y., Poethig R. S. A pathway for the biogenesis of trans-acting siRNAs in Arabidopsis. (англ.) // Genes & development. — 2005. — Vol. 19, no. 18. — P. 2164—2175. — doi:10.1101/gad.1352605. — PMID 16131612.
- ↑ Amaral P. P., Mattick J. S. Noncoding RNA in development. (англ.) // Mammalian genome : official journal of the International Mammalian Genome Society. — 2008. — Vol. 19, no. 7-8. — P. 454—492. — doi:10.1007/s00335-008-9136-7. — PMID 18839252.
- ↑ 1 2 Liu D., Brockman J. M., Dass B., Hutchins L. N., Singh P., McCarrey J. R., MacDonald C. C., Graber J. H. Systematic variation in mRNA 3'-processing signals during mouse spermatogenesis. (англ.) // Nucleic acids research. — 2007. — Vol. 35, no. 1. — P. 234—246. — doi:10.1093/nar/gkl919. — PMID 17158511.
- ↑ Lutz C. S. Alternative polyadenylation: a twist on mRNA 3' end formation. (англ.) // ACS chemical biology. — 2008. — Vol. 3, no. 10. — P. 609—617. — doi:10.1021/cb800138w. — PMID 18817380.
- ↑ 1 2 Beaudoing E., Freier S., Wyatt J. R., Claverie J. M., Gautheret D. Patterns of variant polyadenylation signal usage in human genes. (англ.) // Genome research. — 2000. — Vol. 10, no. 7. — P. 1001—1010. — PMID 10899149.
- ↑ Brown K. M., Gilmartin G. M. A mechanism for the regulation of pre-mRNA 3' processing by human cleavage factor Im. (англ.) // Molecular cell. — 2003. — Vol. 12, no. 6. — P. 1467—1476. — PMID 14690600.
- ↑ Yang Q., Gilmartin G. M., Doublié S. Structural basis of UGUA recognition by the Nudix protein CFI(m)25 and implications for a regulatory role in mRNA 3' processing. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2010. — Vol. 107, no. 22. — P. 10062—10067. — doi:10.1073/pnas.1000848107. — PMID 20479262.
- ↑ Yang Q., Coseno M., Gilmartin G. M., Doublié S. Crystal structure of a human cleavage factor CFI(m)25/CFI(m)68/RNA complex provides an insight into poly(A) site recognition and RNA looping. (англ.) // Structure (London, England : 1993). — 2011. — Vol. 19, no. 3. — P. 368—377. — doi:10.1016/j.str.2010.12.021. — PMID 21295486.
- ↑ Venkataraman K., Brown K. M., Gilmartin G. M. Analysis of a noncanonical poly(A) site reveals a tripartite mechanism for vertebrate poly(A) site recognition. (англ.) // Genes & development. — 2005. — Vol. 19, no. 11. — P. 1315—1327. — doi:10.1101/gad.1298605. — PMID 15937220.
- ↑ 1 2 Millevoi S., Loulergue C., Dettwiler S., Karaa S. Z., Keller W., Antoniou M., Vagner S. An interaction between U2AF 65 and CF I(m) links the splicing and 3' end processing machineries. (англ.) // The EMBO journal. — 2006. — Vol. 25, no. 20. — P. 4854—4864. — doi:10.1038/sj.emboj.7601331. — PMID 17024186.
- ↑ 1 2 3 Shen Y., Ji G., Haas B. J., Wu X., Zheng J., Reese G. J., Li Q. Q. Genome level analysis of rice mRNA 3'-end processing signals and alternative polyadenylation. (англ.) // Nucleic acids research. — 2008. — Vol. 36, no. 9. — P. 3150—3161. — doi:10.1093/nar/gkn158. — PMID 18411206.
- ↑ Glover-Cutter K., Kim S., Espinosa J., Bentley D. L. RNA polymerase II pauses and associates with pre-mRNA processing factors at both ends of genes. (англ.) // Nature structural & molecular biology. — 2008. — Vol. 15, no. 1. — P. 71—78. — doi:10.1038/nsmb1352. — PMID 18157150.
- ↑ Alberts B, Johnson A, Lewis J, et al. Chapter 6: "From DNA to RNA" // Molecular Biology of the Cell. — 4th edition. — New York: Garland Science, 2002.
- ↑ Stumpf G., Domdey H. Dependence of yeast pre-mRNA 3'-end processing on CFT1: a sequence homolog of the mammalian AAUAAA binding factor. (англ.) // Science (New York, N.Y.). — 1996. — Vol. 274, no. 5292. — P. 1517—1520. — PMID 8929410.
- ↑ Iseli C., Stevenson B. J., de Souza S. J., Samaia H. B., Camargo A. A., Buetow K. H., Strausberg R. L., Simpson A. J., Bucher P., Jongeneel C. V. Long-range heterogeneity at the 3' ends of human mRNAs. (англ.) // Genome research. — 2002. — Vol. 12, no. 7. — P. 1068—1074. — doi:10.1101/gr.62002. — PMID 12097343.
- ↑ Balbo P. B., Bohm A. Mechanism of poly(A) polymerase: structure of the enzyme-MgATP-RNA ternary complex and kinetic analysis. (англ.) // Structure (London, England : 1993). — 2007. — Vol. 15, no. 9. — P. 1117—1131. — doi:10.1016/j.str.2007.07.010. — PMID 17850751.
- ↑ Viphakone N., Voisinet-Hakil F., Minvielle-Sebastia L. Molecular dissection of mRNA poly(A) tail length control in yeast. (англ.) // Nucleic acids research. — 2008. — Vol. 36, no. 7. — P. 2418—2433. — doi:10.1093/nar/gkn080. — PMID 18304944.
- ↑ Wahle E. Poly(A) tail length control is caused by termination of processive synthesis. (англ.) // The Journal of biological chemistry. — 1995. — Vol. 270, no. 6. — P. 2800—2808. — PMID 7852352.
- ↑ Dichtl B., Blank D., Sadowski M., Hübner W., Weiser S., Keller W. Yhh1p/Cft1p directly links poly(A) site recognition and RNA polymerase II transcription termination. (англ.) // The EMBO journal. — 2002. — Vol. 21, no. 15. — P. 4125—4135. — PMID 12145212.
- ↑ Nag A., Narsinh K., Martinson H. G. The poly(A)-dependent transcriptional pause is mediated by CPSF acting on the body of the polymerase. (англ.) // Nature structural & molecular biology. — 2007. — Vol. 14, no. 7. — P. 662—669. — doi:10.1038/nsmb1253. — PMID 17572685.
- ↑ Tefferi A., Wieben E. D., Dewald G. W., Whiteman D. A., Bernard M. E., Spelsberg T. C. Primer on medical genomics part II: Background principles and methods in molecular genetics. (англ.) // Mayo Clinic proceedings. — 2002. — Vol. 77, no. 8. — P. 785—808. — doi:10.4065/77.8.785. — PMID 12173714.
- ↑ Coller J. M., Gray N. K., Wickens M. P. mRNA stabilization by poly(A) binding protein is independent of poly(A) and requires translation. (англ.) // Genes & development. — 1998. — Vol. 12, no. 20. — P. 3226—3235. — PMID 9784497.
- ↑ 1 2 Siddiqui N., Mangus D. A., Chang T. C., Palermino J. M., Shyu A. B., Gehring K. Poly(A) nuclease interacts with the C-terminal domain of polyadenylate-binding protein domain from poly(A)-binding protein. (англ.) // The Journal of biological chemistry. — 2007. — Vol. 282, no. 34. — P. 25067—25075. — doi:10.1074/jbc.M701256200. — PMID 17595167.
- ↑ Vinciguerra P., Stutz F. mRNA export: an assembly line from genes to nuclear pores. (англ.) // Current opinion in cell biology. — 2004. — Vol. 16, no. 3. — P. 285—292. — doi:10.1016/j.ceb.2004.03.013. — PMID 15145353.
- ↑ Gray N. K., Coller J. M., Dickson K. S., Wickens M. Multiple portions of poly(A)-binding protein stimulate translation in vivo. (англ.) // The EMBO journal. — 2000. — Vol. 19, no. 17. — P. 4723—4733. — doi:10.1093/emboj/19.17.4723. — PMID 10970864.
- ↑ Спирин, 2011, с. 352—353.
- ↑ Barrett et. al., 2013, p. 29—30.
- ↑ Meaux S., Van Hoof A. Yeast transcripts cleaved by an internal ribozyme provide new insight into the role of the cap and poly(A) tail in translation and mRNA decay. (англ.) // RNA (New York, N.Y.). — 2006. — Vol. 12, no. 7. — P. 1323—1337. — doi:10.1261/rna.46306. — PMID 16714281.
- ↑ 1 2 Meijer H. A., Bushell M., Hill K., Gant T. W., Willis A. E., Jones P., de Moor C. H. A novel method for poly(A) fractionation reveals a large population of mRNAs with a short poly(A) tail in mammalian cells. (англ.) // Nucleic acids research. — 2007. — Vol. 35, no. 19. — P. e132. — doi:10.1093/nar/gkm830. — PMID 17933768.
- ↑ Lehner B., Sanderson C. M. A protein interaction framework for human mRNA degradation. (англ.) // Genome research. — 2004. — Vol. 14, no. 7. — P. 1315—1323. — doi:10.1101/gr.2122004. — PMID 15231747.
- ↑ Wu L., Fan J., Belasco J. G. MicroRNAs direct rapid deadenylation of mRNA. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2006. — Vol. 103, no. 11. — P. 4034—4039. — doi:10.1073/pnas.0510928103. — PMID 16495412.
- ↑ Cui J., Sackton K. L., Horner V. L., Kumar K. E., Wolfner M. F. Wispy, the Drosophila homolog of GLD-2, is required during oogenesis and egg activation. (англ.) // Genetics. — 2008. — Vol. 178, no. 4. — P. 2017—2029. — doi:10.1534/genetics.107.084558. — PMID 18430932.
- ↑ Спирин, 2011, с. 416.
- ↑ Wilusz C. J., Wormington M., Peltz S. W. The cap-to-tail guide to mRNA turnover. (англ.) // Nature reviews. Molecular cell biology. — 2001. — Vol. 2, no. 4. — P. 237—246. — doi:10.1038/35067025. — PMID 11283721.
- ↑ Tian B., Hu J., Zhang H., Lutz C. S. A large-scale analysis of mRNA polyadenylation of human and mouse genes. (англ.) // Nucleic acids research. — 2005. — Vol. 33, no. 1. — P. 201—212. — doi:10.1093/nar/gki158. — PMID 15647503.
- ↑ Danckwardt S., Hentze M. W., Kulozik A. E. 3' end mRNA processing: molecular mechanisms and implications for health and disease. (англ.) // The EMBO journal. — 2008. — Vol. 27, no. 3. — P. 482—498. — doi:10.1038/sj.emboj.7601932. — PMID 18256699.
- ↑ Sandberg R., Neilson J. R., Sarma A., Sharp P. A., Burge C. B. Proliferating cells express mRNAs with shortened 3' untranslated regions and fewer microRNA target sites. (англ.) // Science (New York, N.Y.). — 2008. — Vol. 320, no. 5883. — P. 1643—1647. — doi:10.1126/science.1155390. — PMID 18566288.
- ↑ Tili E., Michaille J. J., Calin G. A. Expression and function of micro-RNAs in immune cells during normal or disease state. (англ.) // International journal of medical sciences. — 2008. — Vol. 5, no. 2. — P. 73—79. — PMID 18392144.
- ↑ Ghosh T., Soni K., Scaria V., Halimani M., Bhattacharjee C., Pillai B. MicroRNA-mediated up-regulation of an alternatively polyadenylated variant of the mouse cytoplasmic {beta}-actin gene. (англ.) // Nucleic acids research. — 2008. — Vol. 36, no. 19. — P. 6318—6332. — doi:10.1093/nar/gkn624. — PMID 18835850.
- ↑ Alt F. W., Bothwell A. L., Knapp M., Siden E., Mather E., Koshland M., Baltimore D. Synthesis of secreted and membrane-bound immunoglobulin mu heavy chains is directed by mRNAs that differ at their 3' ends. (англ.) // Cell. — 1980. — Vol. 20, no. 2. — P. 293—301. — PMID 6771018.
- ↑ Tian B., Pan Z., Lee J. Y. Widespread mRNA polyadenylation events in introns indicate dynamic interplay between polyadenylation and splicing. (англ.) // Genome research. — 2007. — Vol. 17, no. 2. — P. 156—165. — doi:10.1101/gr.5532707. — PMID 17210931.
- ↑ 1 2 Shell S. A., Hesse C., Morris S. M. Jr., Milcarek C. Elevated levels of the 64-kDa cleavage stimulatory factor (CstF-64) in lipopolysaccharide-stimulated macrophages influence gene expression and induce alternative poly(A) site selection. (англ.) // The Journal of biological chemistry. — 2005. — Vol. 280, no. 48. — P. 39950—39961. — doi:10.1074/jbc.M508848200. — PMID 16207706.
- ↑ 1 2 Danckwardt S., Gantzert A. S., Macher-Goeppinger S., Probst H. C., Gentzel M., Wilm M., Gröne H. J., Schirmacher P., Hentze M. W., Kulozik A. E. p38 MAPK controls prothrombin expression by regulated RNA 3' end processing. (англ.) // Molecular cell. — 2011. — Vol. 41, no. 3. — P. 298—310. — doi:10.1016/j.molcel.2010.12.032. — PMID 21292162.
- ↑ Licatalosi D. D., Mele A., Fak J. J., Ule J., Kayikci M., Chi S. W., Clark T. A., Schweitzer A. C., Blume J. E., Wang X., Darnell J. C., Darnell R. B. HITS-CLIP yields genome-wide insights into brain alternative RNA processing. (англ.) // Nature. — 2008. — Vol. 456, no. 7221. — P. 464—469. — doi:10.1038/nature07488. — PMID 18978773.
- ↑ Hall-Pogar T., Liang S., Hague L. K., Lutz C. S. Specific trans-acting proteins interact with auxiliary RNA polyadenylation elements in the COX-2 3'-UTR. (англ.) // RNA (New York, N.Y.). — 2007. — Vol. 13, no. 7. — P. 1103—1115. — doi:10.1261/rna.577707. — PMID 17507659.
- ↑ Danckwardt S., Kaufmann I., Gentzel M., Foerstner K. U., Gantzert A. S., Gehring N. H., Neu-Yilik G., Bork P., Keller W., Wilm M., Hentze M. W., Kulozik A. E. Splicing factors stimulate polyadenylation via USEs at non-canonical 3' end formation signals. (англ.) // The EMBO journal. — 2007. — Vol. 26, no. 11. — P. 2658—2669. — doi:10.1038/sj.emboj.7601699. — PMID 17464285.
- ↑ Wood A. J., Schulz R., Woodfine K., Koltowska K., Beechey C. V., Peters J., Bourc'his D., Oakey R. J. Regulation of alternative polyadenylation by genomic imprinting. (англ.) // Genes & development. — 2008. — Vol. 22, no. 9. — P. 1141—1146. — doi:10.1101/gad.473408. — PMID 18451104.
- ↑ Jung M. Y., Lorenz L., Richter J. D. Translational control by neuroguidin, a eukaryotic initiation factor 4E and CPEB binding protein. (англ.) // Molecular and cellular biology. — 2006. — Vol. 26, no. 11. — P. 4277—4287. — doi:10.1128/MCB.02470-05. — PMID 16705177.
- ↑ Sakurai T., Sato M., Kimura M. Diverse patterns of poly(A) tail elongation and shortening of murine maternal mRNAs from fully grown oocyte to 2-cell embryo stages. (англ.) // Biochemical and biophysical research communications. — 2005. — Vol. 336, no. 4. — P. 1181—1189. — doi:10.1016/j.bbrc.2005.08.250. — PMID 16169522.
- ↑ Taft R. A. Virtues and limitations of the preimplantation mouse embryo as a model system. (англ.) // Theriogenology. — 2008. — Vol. 69, no. 1. — P. 10—16. — doi:10.1016/j.theriogenology.2007.09.032. — PMID 18023855.
- ↑ Richter J. D. CPEB: a life in translation. (англ.) // Trends in biochemical sciences. — 2007. — Vol. 32, no. 6. — P. 279—285. — doi:10.1016/j.tibs.2007.04.004. — PMID 17481902.
- ↑ Piqué M., López J. M., Foissac S., Guigó R., Méndez R. A combinatorial code for CPE-mediated translational control. (англ.) // Cell. — 2008. — Vol. 132, no. 3. — P. 434—448. — doi:10.1016/j.cell.2007.12.038. — PMID 18267074.
- ↑ Benoit P., Papin C., Kwak J. E., Wickens M., Simonelig M. PAP- and GLD-2-type poly(A) polymerases are required sequentially in cytoplasmic polyadenylation and oogenesis in Drosophila. (англ.) // Development (Cambridge, England). — 2008. — Vol. 135, no. 11. — P. 1969—1979. — doi:10.1242/dev.021444. — PMID 18434412.
- ↑ Reinisch K. M., Wolin S. L. Emerging themes in non-coding RNA quality control. (англ.) // Current opinion in structural biology. — 2007. — Vol. 17, no. 2. — P. 209—214. — doi:10.1016/j.sbi.2007.03.012. — PMID 17395456.
- ↑ Jia H., Wang X., Liu F., Guenther U. P., Srinivasan S., Anderson J. T., Jankowsky E. The RNA helicase Mtr4p modulates polyadenylation in the TRAMP complex. (англ.) // Cell. — 2011. — Vol. 145, no. 6. — P. 890—901. — doi:10.1016/j.cell.2011.05.010. — PMID 21663793.
- ↑ 1 2 Martin G., Keller W. RNA-specific ribonucleotidyl transferases. (англ.) // RNA (New York, N.Y.). — 2007. — Vol. 13, no. 11. — P. 1834—1849. — doi:10.1261/rna.652807. — PMID 17872511.
- ↑ Slomovic S., Laufer D., Geiger D., Schuster G. Polyadenylation of ribosomal RNA in human cells. (англ.) // Nucleic acids research. — 2006. — Vol. 34, no. 10. — P. 2966—2975. — doi:10.1093/nar/gkl357. — PMID 16738135.
- ↑ Régnier P., Arraiano C. M. Degradation of mRNA in bacteria: emergence of ubiquitous features. (англ.) // BioEssays : news and reviews in molecular, cellular and developmental biology. — 2000. — Vol. 22, no. 3. — P. 235—244. — doi:10.1002/(SICI)1521-1878(200003)22:3<235::AID-BIES5>3.0.CO;2-2. — PMID 10684583.
- ↑ 1 2 3 Anantharaman V., Koonin E. V., Aravind L. Comparative genomics and evolution of proteins involved in RNA metabolism. (англ.) // Nucleic acids research. — 2002. — Vol. 30, no. 7. — P. 1427—1464. — PMID 11917006.
- ↑ 1 2 Shimyn Slomovic, Victoria Portnoy, Varda Liveanu & Gadi Schuster. RNA Polyadenylation in Prokaryotes and Organelles; Different Tails Tell Different Tales (англ.) // Critical Reviews in Plant Sciences. — 2006. — Т. 25, № 1. — С. 65—77. — doi:10.1080/07352680500391337.
- ↑ Chang S. A., Cozad M., Mackie G. A., Jones G. H. Kinetics of polynucleotide phosphorylase: comparison of enzymes from Streptomyces and Escherichia coli and effects of nucleoside diphosphates. (англ.) // Journal of bacteriology. — 2008. — Vol. 190, no. 1. — P. 98—106. — doi:10.1128/JB.00327-07. — PMID 17965156.
- ↑ Nagaike T., Suzuki T., Ueda T. Polyadenylation in mammalian mitochondria: insights from recent studies. (англ.) // Biochimica et biophysica acta. — 2008. — Vol. 1779, no. 4. — P. 266—269. — doi:10.1016/j.bbagrm.2008.02.001. — PMID 18312863.
- ↑ Walter M., Kilian J., Kudla J. PNPase activity determines the efficiency of mRNA 3'-end processing, the degradation of tRNA and the extent of polyadenylation in chloroplasts. (англ.) // The EMBO journal. — 2002. — Vol. 21, no. 24. — P. 6905—6914. — PMID 12486011.
- ↑ Portnoy V., Schuster G. RNA polyadenylation and degradation in different Archaea; roles of the exosome and RNase R. (англ.) // Nucleic acids research. — 2006. — Vol. 34, no. 20. — P. 5923—5931. — doi:10.1093/nar/gkl763. — PMID 17065466.
- ↑ Yehudai-Resheff S., Portnoy V., Yogev S., Adir N., Schuster G. Domain analysis of the chloroplast polynucleotide phosphorylase reveals discrete functions in RNA degradation, polyadenylation, and sequence homology with exosome proteins. (англ.) // The Plant cell. — 2003. — Vol. 15, no. 9. — P. 2003—2019. — PMID 12953107.
- ↑ Sarkar N. Polyadenylation of mRNA in prokaryotes. (англ.) // Annual review of biochemistry. — 1997. — Vol. 66. — P. 173—197. — doi:10.1146/annurev.biochem.66.1.173. — PMID 9242905.
- ↑ Slomovic S., Portnoy V., Schuster G. Detection and characterization of polyadenylated RNA in Eukarya, Bacteria, Archaea, and organelles. (англ.) // Methods in enzymology. — 2008. — Vol. 447. — P. 501—520. — doi:10.1016/S0076-6879(08)02224-6. — PMID 19161858.
- ↑ Portnoy V., Evguenieva-Hackenberg E., Klein F., Walter P., Lorentzen E., Klug G., Schuster G. RNA polyadenylation in Archaea: not observed in Haloferax while the exosome polynucleotidylates RNA in Sulfolobus. (англ.) // EMBO reports. — 2005. — Vol. 6, no. 12. — P. 1188—1193. — doi:10.1038/sj.embor.7400571. — PMID 16282984.
- ↑ Portnoy V., Schuster G. Mycoplasma gallisepticum as the first analyzed bacterium in which RNA is not polyadenylated. (англ.) // FEMS microbiology letters. — 2008. — Vol. 283, no. 1. — P. 97—103. — doi:10.1111/j.1574-6968.2008.01157.x. — PMID 18399989.
- ↑ Evguenieva-Hackenberg E., Roppelt V., Finsterseifer P., Klug G. Rrp4 and Csl4 are needed for efficient degradation but not for polyadenylation of synthetic and natural RNA by the archaeal exosome. (англ.) // Biochemistry. — 2008. — Vol. 47, no. 50. — P. 13158—13168. — doi:10.1021/bi8012214. — PMID 19053279.
- ↑ 1 2 Shimyn Slomovic, Victoria Portnoy, Shlomit Yehudai-Resheff, Ela Bronshtein, Gadi Schuster. Polynucleotide phosphorylase and the archaeal exosome as poly(A)-polymerases. (англ.) // Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. — 2008. — Т. 1179, № 4. — С. 247—55. — doi:10.1016/j.bbagrm.2007.12.004.
- ↑ EDMONDS M., ABRAMS R. Polynucleotide biosynthesis: formation of a sequence of adenylate units from adenosine triphosphate by an enzyme from thymus nuclei. (англ.) // The Journal of biological chemistry. — 1960. — Vol. 235. — P. 1142—1149. — PMID 13819354.
- ↑ Colgan D. F., Manley J. L. Mechanism and regulation of mRNA polyadenylation. (англ.) // Genes & development. — 1997. — Vol. 11, no. 21. — P. 2755—2766. — PMID 9353246.
- ↑ 1 2 Mary Edmonds. A history of poly A sequences: from formation to factors to function. (англ.) // Progress in Nucleic Acid Research and Molecular Biology. — 2002. — Т. 71. — С. 285—389. — doi:10.1016/S0079-6603(02)71046-5.
- ↑ Mary Edmonds, Maurice H. Vaughan, Jr., Hiroshi Nakazato. Polyadenylic Acid Sequences in the Heterogeneous Nuclear RNA and Rapidly-Labeled Polyribosomal RNA of HeLa Cells: Possible Evidence for a Precursor Relationship (англ.) // Proc Natl Acad Sci U S A.. — 1971. — Т. 68, № 6. — С. 1336—1340. Архивировано 24 сентября 2015 года.
Литература
[править | править код]- Спирин А. С. Молекулярная биология. Рибосомы и биосинтез белка. — М.: Издательский центр «Академия», 2011. — 496 с. — ISBN 978-5-7695-6668-4.
- Lucy W. Barrett, Sue Fletcher, Steve D. Wilton. Untranslated Gene Regions and Other Non-coding Elements. — SpringerBriefs in Biochemistry and Molecular Biology, 2013. — 57 p. — ISBN 978-3-0348-0679-4.
- Elkon R., Ugalde A. P., Agami R. Alternative cleavage and polyadenylation: extent, regulation and function. (англ.) // Nature reviews. Genetics. — 2013. — Vol. 14, no. 7. — P. 496—506. — doi:10.1038/nrg3482. — PMID 23774734.
Ссылки
[править | править код]- Старокадомский, Пётр. мРНКаааауу. // Сайт Biomolecula.ru (30 июня 2009). Дата обращения: 26 марта 2018. Архивировано 18 марта 2018 года.
Эта статья входит в число хороших статей русскоязычного раздела Википедии. |