Трёхцепочечная ДНК (Mj~]eyhkcycugx :UT)
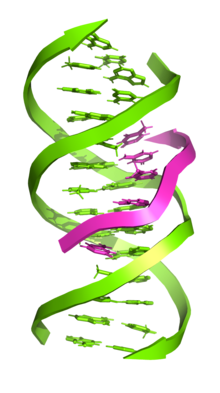
Трёхцепочечная ДНК, H-ДНК или три́плекс-ДНК — форма ДНК, в которой три олигонуклеотида обвивают друг друга, формируя тройную спираль. В трёхцепочечной ДНК третья цепь ДНК связывается с двуцепочечной B-формой ДНК, сформированной за счёт уотсон—криковских взаимодействий, с помощью хугстиновских взаимодействий или обратных хугстиновских водородных связей. Трёхцепочечная ДНК может мешать нормальной репликации и повышать частоту мутаций в области формирования.
Триплекс-формирующими олигонуклеотидами (ТФО) называют олигонуклеотиды длиной 15—25 нуклеотидов, которые связываются с большой бороздкой двухцепочечной ДНК, образуя межмолекулярную триплексную ДНК. ТФО могут подавлять транскрипцию за счёт связывания с двойной спиралью ДНК, поскольку при этом сайты связывания транскрипционных факторов оказываются недоступны. Введение ТФО в клетку может быть использовано для контроля экспрессии генов, сайт-направленного мутагенеза и в перспективе может стать одной из стратегий генной терапии.
Структура
[править | править код]Хугстиновские пары оснований
[править | править код]
Тимин (T) может взаимодействовать с уотсон—криковской парой T—A посредством хугстиновской водородной связи. Тимин формирует водородные связи с аденином в исходной паре T—A с образованием триплета вида T—A*T[1]. В кислой среде протонированный цитозин (C+) может взаимодействовать с парой C—G также посредством хугстиновских взаимодействий, образуя триплет C—G*C+. Триплеты T—A*T и C—G*C+ — это самые стабильные из возможных триплетов, в то время как триплеты T—A*G и C—G*G являются наименее стабильными[2].
Внутримолекулярные и межмолекулярные взаимодействия
[править | править код]
Существует два класса трёхцепочечных ДНК: внутримолекулярные и межмолекулярные. В случае межмолекулярной триплексной ДНК связь образуется между дуплексом ДНК и ещё одной, внешней цепью ДНК, которая может быть как из гомологичной хромосомы, так и являться триплекс-формирующим нуклеотидом (англ. triplex forming oligonucleotide, TFO). Внутримолекулярная трёхцепочечная ДНК формируется из дуплекса с гомопуриновыми и гомопиримидиновыми участками с повторяющейся зеркальной симметрией[3]. На количество сформированной внутримолекулярной трёхцепочечной ДНК влияет степень сверхспирализации ДНК[4]. Выделяют два типа внутримолекулярной триплексной ДНК: H-ДНК и H*-ДНК. H-ДНК формируется в кислой среде в присутствии двухвалентных катионов, таких как Mg2+. В этой конформации гомопиримидиновая цепь в дуплексе выворачивается назад, чтобы связаться параллельным образом с пуриновой цепью. Эту конформацию стабилизируют триады оснований T—A*T и C—G*A+. В случае последней триады цитозин должен быть протонирован, именно поэтому для формирования H-ДНК необходима кислая среда[5]. H*-ДНК образуется при нейтральных значениях pH в присутствии двухвалентных катионов. В случае H*-ДНК гомопиримидиновая и пуриновая цепи связываются друг с другом антипараллельно. H*-ДНК стабилизируется за счет триад T—A*A и C—G*G[3][5].
Образование
[править | править код]Триплекс-формирующие олигонуклеотиды
[править | править код]Триплекс-формирующими олигонуклеотидами (ТФО) называют олигонуклеотиды длиной 15—25 нуклеотидов, которые связываются с большой бороздкой двухцепочечной ДНК, образуя межмолекулярную триплексную ДНК. Получен ряд свидетельств того, что in vivo эти олигонуклеотиды могут принимать участие в регуляции экспрессии генов[6].
ТФО, как правило, связываются с гомопуриновыми или гомопиримидиновыми участками, которые наиболее часто встречаются в области промоторов и интронов в составе генов[7]. ТФО могут подавлять транскрипцию за счёт связывания с двойной спиралью ДНК, поскольку при этом сайты связывания транскрипционных факторов оказываются недоступны. Введение ТФО в клетку с помощью трансфекции или иными способами может быть использовано для контроля экспрессии генов[8], сайт-направленного мутагенеза и в перспективе может стать одной из стратегий генной терапии. Например, в 2004 году был создан ТФО, который специфично связывает промотор гена, кодирующего транскрипционный фактор ETS2[англ.], оверэкспрессия которого часто наблюдается при раке простаты[9]. Разработан ТФО, который специфично взаимодействует с промотором гена bcl-2, кодирующего белок-репрессор апоптоза[10].
Подавление транскрипции в результате образования трёхцепочечной ДНК может лежать в основе заболеваний и патологических состояний человека. Так, при атаксии Фридрейха образование H-ДНК блокирует экспрессию интрона 1 гена FXN[11]. В конечном итоге это приводит к запуску нейродегенеративных процессов в нервной системе и двигательным нарушениям в конечностях[12]. Образование триплексной ДНК может распознаваться системой эксцизионной репарации нуклеотидов, которая восстанавливает двухцепочечную структуру ДНК[13].
Пептидонуклеиновые кислоты
[править | править код]
С дуплексной ДНК также могут взаимодействовать синтетические пептидонуклеиновые кислоты (ПНК), у которых сахарофосфатный остов заменен псевдопептидным скелетом. Когда пептидонуклеиновая кислота связывается с дуплексной ДНК, то одна из цепей ДНК вытесняется, и образуется P-петля. Пептидонуклеиновые кислоты устойчивы к действию протеаз и могут быть использованы для запуска репарации в локусе-мишени из-за формирования H-ДНК. ПНК могут связываться с комплементарной цепочкой ДНК с высоким сродством и специфичностью посредством уотсон—криковских взаимодействий. При формировании триплексной ДНК ПНК взаимодействует с дуплексом посредством хугстиновских взаимодействий[14]. В отличие от настоящей триплексной ДНК, гибрид ПНК и двуцепочечной ДНК стабилен, поскольку в состав ПНК входит не отрицательно заряженный сахарофосфатный остов, а нейтральный псевдопептидный скелет[15]. В отличие от ТФО, которые связываются с дуплексной ДНК в области большой бороздки, ПНК взаимодействуют с двойной спиралью ДНК по-разному[14].
Последовательность дуплексной ДНК смешанного состава может быть распознана парой псевдокомплементарных ПНК, которые могут осуществлять двойную инвазию в спираль ДНК за счет одновременного образования диаминопурина[англ.] (D) и тиоурацила[англ.] (US), которые заменяют аденин и тимин соответственно[16]. Псевдокомплементарные ПНК образуют спирали состава ПНК:ДНК с каждой из цепей дуплекса за счёт образования пар D—T, US—A, G—C и С—G. Другую форма инвазии дуплекса может осуществлять гомопуриновая ПНК за счёт комплементарного взаимодействия с антипараллельной цепью ДНК[17][15].
Наконец, ПНК могут образовывать «зажим» (англ. clamp) на сайте-мишени в результате химической модификации. Один вид «зажима» включает структуру из двух ПНК, соединённых гибким линкером — 8-амино-3,6-диоксаоктаноевой кислотой[18]. Эта структура образует триплекс состава ПНК:ДНК:ПНК в сайте-мишени, причем одна из ПНК взаимодействует с антипараллельной цепью ДНК с помощью уотсон-криковских пар, а другая цепь формирует хугстиновские пары с другой цепью, причем вторая цепь обязательно должна содержать гомопуриновый или гомопиримидиновый участок, с которым взаимодействует ПНК[17]. Другая форма «зажима» известна как «хвостовой зажим» (англ. tail clamp). Она состоит из «зажима» ПНК:ДНК:ПНК и дополнительного дуплекса ДНК:ПНК, который формирует «хвост» длиной 5—10 пар оснований. В этом случае необходимости в гомопуриновом или гомопиримидиновом участке в исходном дуплексе ДНК нет[15].
Функции
[править | править код]Формирование такой нестабильной структуры, как H-ДНК, может стать причиной геномной нестабильности в месте ее появления[19]. Например, рядом с промотором P1 гена c-MYC присутствуют полипуриновые участки, способные к формированию триплексной ДНК, причем её образование приводит к повышению нестабильности вблизи c-MYC. Эксперименты на трансгенных мышах[англ.] показали, что внедрение человеческих последовательностей, склонных к переходу в триплексное состояние, вместе с последовательностями, которые могут перейти в Z-ДНК, в участки генома, для которых не было известно случаев генетической нестабильности, приводит к появлению нестабильности в них[20]. Кроме того, известно, что формирование H-ДНК может способствовать транслокациям между 14 и 18 хромосомами, которые лежат в основе многих видов рака, например, фолликулярной лимфомы. Учёные показали, что снижение вероятности формирования H-ДНК снижает и вероятность транслокаций[20][21].
Трёхцепочечная ДНК может мешать нормальной репликации и, как и другие неканонические структуры ДНК, повышать частоту мутаций в области формирования[22]. Как отмечалось выше, H-ДНК может представлять собой физическое препятствие для транскрипции. Как показали эксперименты с РНК-полимеразой T7[англ.], транскрипционный аппарат не мог преодолеть уотсон-криковские и хугстиновские взаимодействия, связывающие цепи триплекса, что приводило к остановке транскрипции[23]. Остановка транскрипции при столкновении транскрипционной машинерии с H-ДНК активирует сопряжённую с транскрипцией репарацию, из-за действия которой H-ДНК вырезается, приводя к делециям[24].
Трёхцепочечная ДНК может распознаваться разными нуклеазами. Например, нуклеазы ERCC1-XPF[англ.] и ERCC1-XPG, задействованные в эксцизионной репарации нуклеотидов, разрезают H-ДНК в области петли, формируемой цепями, которые взаимодействуют при помощи хугстиновских пар, и 5'-концом цепи, которая формирует уотсон-криковские пары[25]. Этот разрыв может приводить к крупным делециям, которые приводят к геномной нестабильности. Нуклеаза FEN1[англ.], напротив, препятствует геномной нестабильности. Она, как и ERCC1-XPG, вносит разрыв в H-ДНК на 5'-конце цепи, не участвующей в хугстиновских взаимодействиях. В клетках линии[англ.] HeLa, лишённых FEN1, количество делеций вблизи H-ДНК больше, чем у клеток с FEN1, причем мутагенный эффект H-ДНК в клетках, лишенных FEN1, был наиболее выражен при репликации ДНК. Таким образом, FEN1 подавляет мутагенез, индуцированный H-ДНК, зависимо от репликации[22][25].
Применение
[править | править код]Как упоминалось выше, ТФО может стать средством генной терапии. Основную сложность в медицинском применении ТФО и пептидонуклеиновых кислот составляет их доставка в клетки[26]. В 2013 году в рамках исследования, нацеленного на изменение экспрессии генов в гемопоэтических клетках, было предложено сшивать молекулы пептидонуклеиновых кислот с пептидом, проникающим в клетки[англ.] (англ. cell penetrating peptide, CPPs), а также наночастицами из поли(молочно-ко-гликоевой кислоты)[англ.][27]. С помощью такого подхода авторам работы удалось модифицировать 6 пар оснований в составе гена CCR5, мутации в котором связывают с устойчивостью к ВИЧ[28]. CCPs позволяют беспрепятственно доставлять внутрь клеток небольшие «грузы», такие как небольшие биомолекулы. Поли(молочно-ко-гликоевая кислота) представляет собой биоразлагаемый полимер, в частицы из которого можно заключить молекулы пептидонуклеиновых кислот. Доставка пептидонуклеиновых кислот в составе наночастиц в клетки-мишени была использована в другом исследовании, посвящённом терапии муковисцидоза. Пептидонуклеиновые кислоты вместе с молекулой ДНК-донором были упакованы в наночастицы и доставлены в клетки эпителия бронхов с целью редактирования мутации в гене CFTR[29].
История изучения
[править | править код]В 1950-х годах, когда точная структура ДНК была неизвестна, трёхцепочечная спираль рассматривалась в качестве одной из возможных моделей организации ДНК в клетке. Трёхцепочечную структуру ДНК как наиболее вероятную рассматривали Лайнус Полинг и Роберт Корей[англ.] (англ. Robert Corey), представившие трёхцепочечную модель ДНК в 1953 году[30][31]. Уотсон и Крик, впоследствии получившие Нобелевскую премию по химии за определение структуры ДНК, также первоначально склонялись к трёхцепочечной модели, однако они увидели в ней ряд проблем, не согласующихся с имеющимися на тот момент экспериментальными данными. В частности, отрицательно заряженные фосфаты, обращённые к оси спирали, отталкивались бы друг от друга в силу электростатических причин, что делало бы невозможным существование стабильной тройной спирали. В трёхцепочечной модели Полинга и Корей некоторые ван-дер-Ваальсовы расстояния были слишком малы. Р. Фрэйзер (англ. R. Fraser) также предложил свою модель тройной спирали, в которой фосфаты были расположены на поверхности спирали, а внутрь были обращены азотистые основания, однако она не подкреплялась экспериментальными данными[32].
Альтернативная структура трёхцепочечной ДНК была предложена в 1957 году[33]. Дж. Фензенфельд, Д. Р. Дэвис и А. Рич предсказали, что стабильная трёхцепочечная ДНК может быть сформирована, если одна цепь состоит только из пуриновых нуклеотидов, а две другие — только из пиримидинов[6][33]. Считалось, что in vivo такая структура формируется только как промежуточный продукт действия белка RecA[англ.] в ходе рекомбинации у бактерии Escherichia coli. Модели трёхцепочечной ДНК, предложенные в 1960-х годах, предсказывали формирование не уотсон-криковских, а хугстиновских пар в большой бороздке ДНК[6]. Спустя некоторое время была предложена модель трёхцепочечной ДНК, состоящей из одной пиримидиновой и двух пуриновых цепей[6]. Открытие трёхцепочечной ДНК в живых клетках в конце 1980-х годов в составе суперскрученных плазмид показало, что образование H-ДНК в принципе возможно в живых клетках[34]. Кроме того, вскоре было показано, что гомопиримидиновые и некоторые обогащённые пуринами олигонуклеотиды могут связываться со специфическими последовательностями в дуплексной ДНК, приводя к образованию трёхцепочечной ДНК[35].
Примечания
[править | править код]- ↑ Rhee S., Han Zj., Liu K., Miles H. T., Davies D. R. Structure of a triple helical DNA with a triplex-duplex junction. (англ.) // Biochemistry. — 1999. — 21 December (vol. 38, no. 51). — P. 16810—16815. — doi:10.1021/bi991811m. — PMID 10606513.
- ↑ Mergny J. L., Sun J. S., Rougée M., Montenay-Garestier T., Barcelo F., Chomilier J., Hélène C. Sequence specificity in triple-helix formation: experimental and theoretical studies of the effect of mismatches on triplex stability. (англ.) // Biochemistry. — 1991. — 8 October (vol. 30, no. 40). — P. 9791—9798. — doi:10.1021/bi00104a031. — PMID 1911764.
- ↑ 1 2 Ussery D. W., Sinden R. R. Environmental influences on the in vivo level of intramolecular triplex DNA in Escherichia coli. (англ.) // Biochemistry. — 1993. — 22 June (vol. 32, no. 24). — P. 6206—6213. — doi:10.1021/bi00075a013. — PMID 8512930.
- ↑ Dayn A., Samadashwily G. M., Mirkin S. M. Intramolecular DNA triplexes: unusual sequence requirements and influence on DNA polymerization. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1992. — 1 December (vol. 89, no. 23). — P. 11406—11410. — doi:10.1073/pnas.89.23.11406. — PMID 1454828.
- ↑ 1 2 Lyamichev V. I., Mirkin S. M., Frank-Kamenetskii M. D. Structures of homopurine-homopyrimidine tract in superhelical DNA. (англ.) // Journal Of Biomolecular Structure & Dynamics. — 1986. — February (vol. 3, no. 4). — P. 667—669. — doi:10.1080/07391102.1986.10508454. — PMID 3271043.
- ↑ 1 2 3 4 Frank-Kamenetskii M. D., Mirkin S. M. Triplex DNA structures. (англ.) // Annual Review Of Biochemistry. — 1995. — Vol. 64. — P. 65—95. — doi:10.1146/annurev.bi.64.070195.000433. — PMID 7574496.
- ↑ Brázdová M., Tichý V., Helma R., Bažantová P., Polášková A., Krejčí A., Petr M., Navrátilová L., Tichá O., Nejedlý K., Bennink M. L., Subramaniam V., Bábková Z., Martínek T., Lexa M., Adámik M. p53 Specifically Binds Triplex DNA In Vitro and in Cells. (англ.) // PloS One. — 2016. — Vol. 11, no. 12. — P. e0167439—0167439. — doi:10.1371/journal.pone.0167439. — PMID 27907175.
- ↑ Graham M. K., Brown T. R., Miller P. S. Targeting the human androgen receptor gene with platinated triplex-forming oligonucleotides. (англ.) // Biochemistry. — 2015. — 7 April (vol. 54, no. 13). — P. 2270—2282. — doi:10.1021/bi501565n. — PMID 25768916.
- ↑ Carbone G. M., Napoli S., Valentini A., Cavalli F., Watson D. K., Catapano C. V. Triplex DNA-mediated downregulation of Ets2 expression results in growth inhibition and apoptosis in human prostate cancer cells. (англ.) // Nucleic Acids Research. — 2004. — Vol. 32, no. 14. — P. 4358—4367. — doi:10.1093/nar/gkh744. — PMID 15314206.
- ↑ Shen C., Rattat D., Buck A., Mehrke G., Polat B., Ribbert H., Schirrmeister H., Mahren B., Matuschek C., Reske S. N. Targeting bcl-2 by triplex-forming oligonucleotide--a promising carrier for gene-radiotherapy. (англ.) // Cancer Biotherapy & Radiopharmaceuticals. — 2003. — February (vol. 18, no. 1). — P. 17—26. — doi:10.1089/108497803321269296. — PMID 12667305.
- ↑ Sakamoto N., Chastain P. D., Parniewski P., Ohshima K., Pandolfo M., Griffith J. D., Wells R. D. Sticky DNA: self-association properties of long GAA.TTC repeats in R.R.Y triplex structures from Friedreich's ataxia. (англ.) // Molecular Cell. — 1999. — April (vol. 3, no. 4). — P. 465—475. — doi:10.1016/s1097-2765(00)80474-8. — PMID 10230399.
- ↑ Bacolla A., Wells R. D. Non-B DNA conformations as determinants of mutagenesis and human disease. (англ.) // Molecular Carcinogenesis. — 2009. — April (vol. 48, no. 4). — P. 273—285. — doi:10.1002/mc.20507. — PMID 19306308.
- ↑ Kaushik Tiwari M., Adaku N., Peart N., Rogers F. A. Triplex structures induce DNA double strand breaks via replication fork collapse in NER deficient cells. (англ.) // Nucleic Acids Research. — 2016. — 19 September (vol. 44, no. 16). — P. 7742—7754. — doi:10.1093/nar/gkw515. — PMID 27298253.
- ↑ 1 2 Jain A., Wang G., Vasquez K. M. DNA triple helices: biological consequences and therapeutic potential. (англ.) // Biochimie. — 2008. — August (vol. 90, no. 8). — P. 1117—1130. — doi:10.1016/j.biochi.2008.02.011. — PMID 18331847.
- ↑ 1 2 3 Hansen M. E., Bentin T., Nielsen P. E. High-affinity triplex targeting of double stranded DNA using chemically modified peptide nucleic acid oligomers. (англ.) // Nucleic Acids Research. — 2009. — July (vol. 37, no. 13). — P. 4498—4507. — doi:10.1093/nar/gkp437. — PMID 19474349.
- ↑ Ricciardi A. S., McNeer N. A., Anandalingam K. K., Saltzman W. M., Glazer P. M. Targeted genome modification via triple helix formation. (англ.) // Methods In Molecular Biology (Clifton, N.J.). — 2014. — Vol. 1176. — P. 89—106. — doi:10.1007/978-1-4939-0992-6_8. — PMID 25030921.
- ↑ 1 2 Rogers F. A., Vasquez K. M., Egholm M., Glazer P. M. Site-directed recombination via bifunctional PNA-DNA conjugates. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2002. — 24 December (vol. 99, no. 26). — P. 16695—16700. — doi:10.1073/pnas.262556899. — PMID 12461167.
- ↑ Montazersaheb S., Hejazi M. S., Nozad Charoudeh H. Potential of Peptide Nucleic Acids in Future Therapeutic Applications. (англ.) // Advanced Pharmaceutical Bulletin. — 2018. — November (vol. 8, no. 4). — P. 551—563. — PMID 30607328.
- ↑ McKinney J. A., Wang G., Mukherjee A., Christensen L., Subramanian SHS, Zhao J., Vasquez K. M. Distinct DNA repair pathways cause genomic instability at alternative DNA structures. (англ.) // Nature Communications. — 2020. — 13 January (vol. 11, no. 1). — P. 236—236. — doi:10.1038/s41467-019-13878-9. — PMID 31932649.
- ↑ 1 2 Wang G., Vasquez K. M. Impact of alternative DNA structures on DNA damage, DNA repair, and genetic instability. (англ.) // DNA Repair. — 2014. — July (vol. 19). — P. 143—151. — doi:10.1016/j.dnarep.2014.03.017. — PMID 24767258.
- ↑ Raghavan S. C., Chastain P., Lee J. S., Hegde B. G., Houston S., Langen R., Hsieh C. L., Haworth I. S., Lieber M. R. Evidence for a triplex DNA conformation at the bcl-2 major breakpoint region of the t(14;18) translocation. (англ.) // The Journal Of Biological Chemistry. — 2005. — 17 June (vol. 280, no. 24). — P. 22749—22760. — doi:10.1074/jbc.M502952200. — PMID 15840562.
- ↑ 1 2 Wang G., Vasquez K. M. Effects of Replication and Transcription on DNA Structure-Related Genetic Instability. (англ.) // Genes. — 2017. — 5 January (vol. 8, no. 1). — doi:10.3390/genes8010017. — PMID 28067787.
- ↑ Pandey S., Ogloblina A. M., Belotserkovskii B. P., Dolinnaya N. G., Yakubovskaya M. G., Mirkin S. M., Hanawalt P. C. Transcription blockage by stable H-DNA analogs in vitro. (англ.) // Nucleic Acids Research. — 2015. — 18 August (vol. 43, no. 14). — P. 6994—7004. — doi:10.1093/nar/gkv622. — PMID 26101261.
- ↑ Belotserkovskii B. P., De Silva E., Tornaletti S., Wang G., Vasquez K. M., Hanawalt P. C. A triplex-forming sequence from the human c-MYC promoter interferes with DNA transcription. (англ.) // The Journal Of Biological Chemistry. — 2007. — 2 November (vol. 282, no. 44). — P. 32433—32441. — doi:10.1074/jbc.M704618200. — PMID 17785457.
- ↑ 1 2 Zhao J., Wang G., Del Mundo I. M., McKinney J. A., Lu X., Bacolla A., Boulware S. B., Zhang C., Zhang H., Ren P., Freudenreich C. H., Vasquez K. M. Distinct Mechanisms of Nuclease-Directed DNA-Structure-Induced Genetic Instability in Cancer Genomes. (англ.) // Cell Reports. — 2018. — 30 January (vol. 22, no. 5). — P. 1200—1210. — doi:10.1016/j.celrep.2018.01.014. — PMID 29386108.
- ↑ Hnedzko D., Cheruiyot S. K., Rozners E. Using triple-helix-forming Peptide nucleic acids for sequence-selective recognition of double-stranded RNA. (англ.) // Current Protocols In Nucleic Acid Chemistry. — 2014. — 8 September (vol. 58). — P. 4—60. — doi:10.1002/0471142700.nc0460s58. — PMID 25199637.
- ↑ McNeer N. A., Schleifman E. B., Cuthbert A., Brehm M., Jackson A., Cheng C., Anandalingam K., Kumar P., Shultz L. D., Greiner D. L., Mark Saltzman W., Glazer P. M. Systemic delivery of triplex-forming PNA and donor DNA by nanoparticles mediates site-specific genome editing of human hematopoietic cells in vivo. (англ.) // Gene Therapy. — 2013. — June (vol. 20, no. 6). — P. 658—669. — doi:10.1038/gt.2012.82. — PMID 23076379.
- ↑ Schleifman E. B., Bindra R., Leif J., del Campo J., Rogers F. A., Uchil P., Kutsch O., Shultz L. D., Kumar P., Greiner D. L., Glazer P. M. Targeted disruption of the CCR5 gene in human hematopoietic stem cells stimulated by peptide nucleic acids. (англ.) // Chemistry & Biology. — 2011. — 23 September (vol. 18, no. 9). — P. 1189—1198. — doi:10.1016/j.chembiol.2011.07.010. — PMID 21944757.
- ↑ McNeer N. A., Anandalingam K., Fields R. J., Caputo C., Kopic S., Gupta A., Quijano E., Polikoff L., Kong Y., Bahal R., Geibel J. P., Glazer P. M., Saltzman W. M., Egan M. E. Nanoparticles that deliver triplex-forming peptide nucleic acid molecules correct F508del CFTR in airway epithelium. (англ.) // Nature Communications. — 2015. — 27 April (vol. 6). — P. 6952—6952. — doi:10.1038/ncomms7952. — PMID 25914116.
- ↑ Pauling L., Corey R. B. A Proposed Structure For The Nucleic Acids. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1953. — February (vol. 39, no. 2). — P. 84—97. — doi:10.1073/pnas.39.2.84. — PMID 16578429.
- ↑ PAULING L, COREY RB. Structure of the nucleic acids. (англ.) // Nature. — 1953. — 21 February (vol. 171, no. 4347). — P. 346—346. — doi:10.1038/171346a0. — PMID 13036888.
- ↑ Fraser R. D. The structure of deoxyribose nucleic acid. (англ.) // Journal Of Structural Biology. — 2004. — March (vol. 145, no. 3). — P. 184—185. — doi:10.1016/j.jsb.2004.01.001. — PMID 14997898.
- ↑ 1 2 Felsenfeld G., Davies David R., Rich Alexander. FORMATION OF A THREE-STRANDED POLYNUCLEOTIDE MOLECULE (англ.) // Journal of the American Chemical Society. — 1957. — April (vol. 79, no. 8). — P. 2023—2024. — ISSN 0002-7863. — doi:10.1021/ja01565a074.
- ↑ Hanvey J. C., Shimizu M., Wells R. D. Intramolecular DNA triplexes in supercoiled plasmids. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1988. — September (vol. 85, no. 17). — P. 6292—6296. — doi:10.1073/pnas.85.17.6292. — PMID 3413097.
- ↑ Mirkin S. M., Lyamichev V. I., Drushlyak K. N., Dobrynin V. N., Filippov S. A., Frank-Kamenetskii M. D. DNA H form requires a homopurine-homopyrimidine mirror repeat. (англ.) // Nature. — 1987. — 3 December (vol. 330, no. 6147). — P. 495—497. — doi:10.1038/330495a0. — PMID 2825028.
Эта статья входит в число хороших статей русскоязычного раздела Википедии. |