Herelleviridae (Herelleviridae)
| Herelleviridae | ||||||
|---|---|---|---|---|---|---|
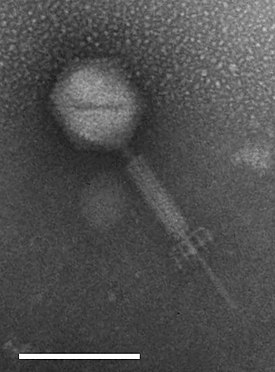 Бактериофаг Schiekvirus EfV12, масштабная черта — 100 нм | ||||||
| Научная классификация | ||||||
|
Реалм: Царство: Тип: Uroviricota Класс: Caudoviricetes Семейство: Herelleviridae |
||||||
| Международное научное название | ||||||
| Herelleviridae | ||||||
| Группа по Балтимору | ||||||
| I: дцДНК-вирусы | ||||||
| ||||||
Herelleviridae — семейство вирулентных бактериофагов с миовирусной морфологией.
История
[править | править код]В 2000-х годах развитие методов геномного анализа позволило выявить гетерогенность классических семейств бактериофагов, выделенных на основе морфологических признаков вирусных частиц[2][3]. В 2009 году Роб Лавин и Эндрю Кропински с соавторами осуществили кластеризацию белковых последовательностей бактериофагов с миовирусной морфологией и предложили подсемейство Spounavirinae в составе семейства Myoviridae. На основании результатов комплексного анализа геномных последовательностей, в 2020 году коллектив авторов в составе Якуба Барыльского, Эвелин Адриансенс и 19 других членов Международного комитета по таксономии вирусов описал сходное по составу семейство Herelleviridae. Названное в честь столетней годовщины открытия вирусов прокариот Феликсом д’Эреллем, оно стало вторым после Ackermannviridae семейством бактериофагов, описанным по результатам биоинформационного анализа общих генов[4][5].
Характеристика
[править | править код]В 2021 году семейство Herelleviridae включало в себя 34 рода бактериофагов. Из них наиболее объёмными были Kayvirus и Pecentumvirus c 11 видами в каждом, а также Bequatrovirus, Caeruleovirus, Harbinvirus и Wphvirus с пятью-шестью видами[6]. Размер капсидов составляет 78—100 нм. Белок хвостовой оболочки состоит из двух или трёх доменов[7]. Размер геномов варьирует в пределах 102—167 т. п. н.[8]. Концы геномов организованы в виде длинных повторов протяжённостью 2—16 т. п. н. Репликация осуществляется при помощи кодируемой геномом фага ДНК-полимеразы. Хозяевами являются бактерии типа Bacillota[9]. Продолжительность латентного периода в жизненном цикле может составлять от 10 до 50 минут[10][11][12]. Представители семейства способны реплицироваться в среднем на 70% бактериальных изолятов, относящихся к виду штамма-продуцента[13]. Диапазон действия у представителей Kayvirus, инфицирующих Staphylococcus aureus, — от 60% до более чем 95% протестированных изолятов[14]. Schiekvirus EfV12 является примером бактериофага, способного размножаться на культурах двух различных видов энтерококков[15].
Практическое значение
[править | править код]В 1997—2014 годах представители родов Kayvirus, Kochikohdavirus и Schiekvirus входили в состав препарата Пиобактериофаг института имени Георга Элиавы[16].
Примечания
[править | править код]- ↑ Таксономия вирусов (англ.) на сайте Международного комитета по таксономии вирусов (ICTV).
- ↑ Turner D; Kropinski AM; Adriaenssens EM (2021). "A roadmap for genome-based phage taxonomy". Viruses. 13 (3): 506. doi:10.3390/v13030506. PMID 33803862.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ Савич В. В., Сидоренко А. В. Некоторые аспекты современной систематики бактериофагов // Микробные биотехнологии: фундаментальные и прикладные аспекты : Сборник научных трудов. — Минск: Республиканское унитарное предприятие "Издательский дом "Белорусская наука", 2021. — Т. 13. — С. 83–102. — doi:10.47612/2226-3136-2021-13-83-102.
- ↑ Lavigne R; Darius P; Summer EJ; Seto D; Mahadevan P; Nilsson AS; Ackermann HW; Kropinski AM (2009). "Classification of Myoviridae bacteriophages using protein sequence similarity". BMC Microbiology. 9: 224. doi:10.1186/1471-2180-9-224. PMID 19857251.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ Virus Taxonomy: 2021 Release. International Committee on Taxonomy of Viruses (ICTV). Дата обращения: 18 января 2023. Архивировано 20 марта 2020 года.
- ↑ Evseev P; Shneider M; Miroshnikov K (2022). "Evolution of phage tail sheath protein". Viruses. 14 (6): 1148. doi:10.3390/v14061148. PMID 35746620.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ [https://www.ncbi.nlm.nih.gov/nuccore/?term=txid2560065[Organism:exp] Nucleotide Database]. National Library of Medicine. Дата обращения: 2023-18-01.
- ↑ Barylski J; Kropinski AM; Alikhan NF; Adriaenssens EM; Ictv Report Consortium (2020). "ICTV Virus Taxonomy Profile: Herelleviridae". The Journal of General Virology. 101 (4): 362—363. doi:10.1099/jgv.0.001392. PMID 32022658.
- ↑ Arroyo-Moreno S; Buttimer C; Bottacini F; Chanishvili N; Ross P; Hill C; Coffey A (2022). "Insights into gene transcriptional regulation of Kayvirus bacteriophages obtained from therapeutic mixtures". Viruses. 14 (3): 626. doi:10.3390/v14030626. PMID 35337034.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ Liu S; Hon K; Bouras GS; Psaltis AJ; Shearwin K; Wormald PJ; Vreugde S (2022). "APTC-C-SA01: A novel bacteriophage cocktail targeting Staphylococcus aureus and MRSA biofilms". International Journal of Molecular Sciences. 23 (11): 6116. doi:10.3390/ijms23116116. PMID 35682794.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ Tkachev PV; Pchelin IM; Azarov DV; Gorshkov AN; Shamova OV; Dmitriev AV; Goncharov AE (2022). "Two novel lytic bacteriophages infecting Enterococcus spp. are promising candidates for targeted antibacterial therapy". Viruses. 14 (4): 831. doi:10.3390/v14040831. PMID 35458561.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ Pchelin IM; Smolensky AV; Azarov DV; Goncharov AE (2024). "Lytic spectra of tailed bacteriophages: a systematic review and meta-analysis". Viruses. 16: 1879. doi:10.3390/v16121879.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ McCallin S; Sarker SA; Sultana S; Oechslin F; Brussow H (2018). "Metagenome analysis of Russian and Georgian Pyophage cocktails and a placebo-controlled safety trial of single phage versus phage cocktail in healthy Staphylococcus aureus carriers". Environmental Microbiology. 20 (9): 3278—3293. doi:10.1111/1462-2920.14310. PMID 30051571.
- ↑ Wandro S; Oliver A; Gallagher T; Weihe C; England W; Martiny JBH; Whiteson K (2019). "Predictable molecular adaptation of coevolving Enterococcus faecium and lytic phage EfV12-phi1". Frontiers in Microbiology. 9: 3192. doi:10.3389/fmicb.2018.03192. PMID 30766528.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ Villarroel J; Larsen MV; Kilstrup M; Nielsen M (2017). "Metagenomic analysis of therapeutic PYO phage cocktails from 1997 to 2014". Viruses. 9 (11): 328. doi:10.3390/v9110328. PMID 29099783.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка)